CDC37 (cell division cycle 37, HSP90 cochaperone)
- symbol:
- CDC37
- locus group:
- protein-coding gene
- location:
- 19p13.2
- gene_family:
- alias symbol:
- P50CDC37
- alias name:
- CDC37 cell division cycle 37 homol…
- entrez id:
- 11140
- ensembl gene id:
- ENSG00000105401
- ucsc gene id:
- uc002mof.2
- refseq accession:
- NM_007065
- hgnc_id:
- HGNC:1735
- approved reserved:
- 1999-09-07
CDC37是一种重要的分子伴侣蛋白,属于热休克蛋白(HSP)辅助伴侣家族,主要功能是帮助稳定和激活激酶类蛋白(如蛋白激酶),尤其是那些依赖热休克蛋白90(HSP90)的激酶。CDC37通过与HSP90形成复合物,协助新生或未折叠的激酶正确折叠并维持其稳定性,从而确保这些激酶能够正常发挥功能。CDC37的作用位点主要在细胞质中,参与多种信号通路的调控,包括细胞增殖、存活和应激反应等。如果CDC37发生突变,可能导致其与HSP90或激酶的结合能力下降,进而影响激酶的稳定性和功能,最终可能引发细胞周期紊乱、凋亡异常或肿瘤发生。研究表明,CDC37的异常表达与多种癌症相关,如乳腺癌、前列腺癌和结直肠癌,其过表达可能通过过度激活致癌激酶(如AKT、ERK等)促进肿瘤生长和转移;而降低CDC37表达则可能导致依赖它的激酶失活,影响细胞正常功能甚至引发细胞死亡。CDC37属于HSP辅助伴侣家族,该家族成员通常通过与其他HSP(如HSP70、HSP90)协作,参与蛋白质折叠、稳定性和功能调控。家族共性包括在应激条件下高表达,协助维持蛋白质稳态(proteostasis),并在细胞保护、信号转导和疾病发展中起关键作用。CDC37的过表达可能扰乱激酶网络的平衡,导致信号通路过度激活;而表达不足则可能影响激酶依赖性生理过程,如免疫响应或代谢调控。
The protein encoded by this gene is highly similar to Cdc 37, a cell division cycle control protein of Sacchromyces cerevisiae. This protein is a molecular chaperone with specific function in cell signal transduction. It has been shown to form complex with Hsp90 and a variety of protein kinases including CDK4, CDK6, SRC, RAF-1, MOK, as well as eIF2 alpha kinases. It is thought to play a critical role in directing Hsp90 to its target kinases. [provided by RefSeq, Jul 2008]
由该基因编码的蛋白质是高度相似疾病预防控制中心37中,的Sacchromyces酵母的细胞分裂周期调控蛋白。这种蛋白是分子伴侣在细胞信号转导特定的功能。已经表明,以形成具有的Hsp90复杂和多种蛋白激酶包括CDK4,CDK6,SRC,RAF-1,莫,以及的eIF2的α激酶。它被认为是在指导的Hsp90与其靶激酶发挥重要的作用。 [由RefSeq的,2008年7月提供]
基因本体信息
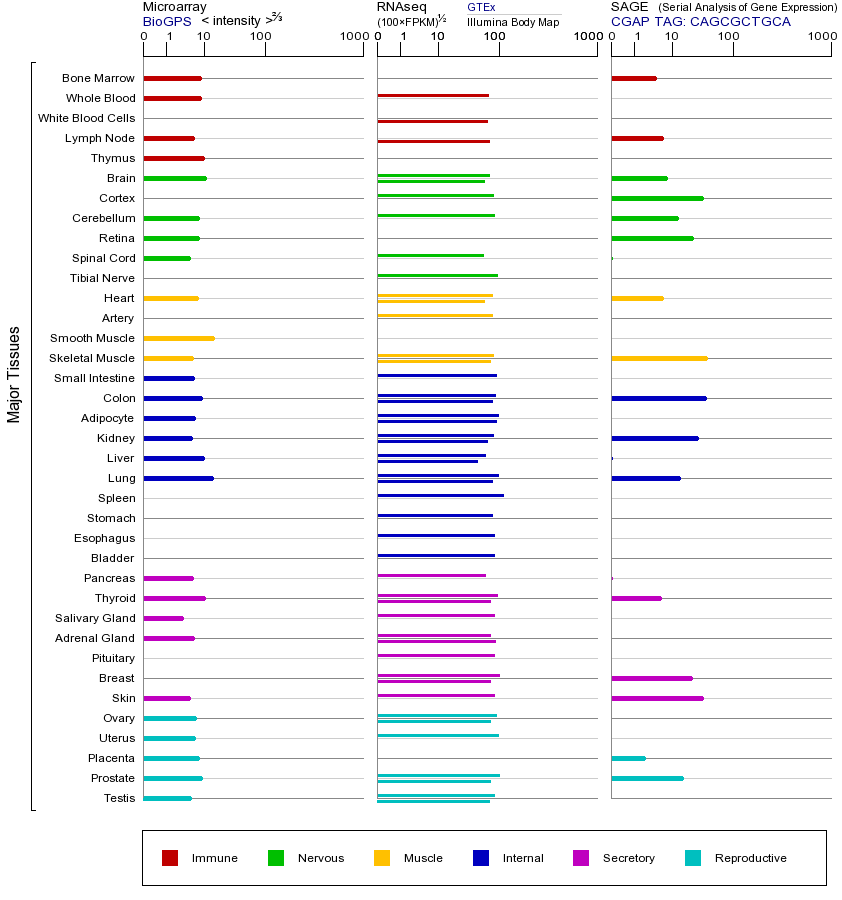
CDC37基因(以及对应的蛋白质)的细胞分布位置:
- 质膜
- 细胞质
- 细胞外
- 高尔基体
- 囊泡
- 细胞骨架
- 内质网
- 细胞核
- 内体
- 溶酶体
- 线粒体
CDC37基因的本体(GO)信息:
| 名称 |
|---|
| 4151 PI3K-Akt signaling pathway [PATH:hsa04151] |
| 名称 |
|---|
| Constitutive Signaling by EGFRvIII |
| Constitutive Signaling by Ligand-Responsive EGFR Cancer Variants |
| Disease |
| Diseases of signal transduction |
| Signal Transduction |
| Signaling by EGFR in Cancer |
| Signaling by EGFRvIII in Cancer |
| Signaling by ERBB2 |
| Signaling by Ligand-Responsive EGFR Variants in Cancer |
| 疾病名称 | 关系值 | NofPmids | NofSnps | 来源 |
| Inflammatory Bowel Diseases | 0.12 | 1 | 1 | GWASCAT |
| Prostate carcinoma | 0.000814326 | 3 | 0 | BeFree |
| Malignant neoplasm of prostate | 0.000814326 | 3 | 0 | BeFree |
| Multiple Myeloma | 0.000542884 | 2 | 0 | BeFree |
| Liver carcinoma | 0.000542884 | 2 | 0 | BeFree |
| invasive aspergillosis | 0.000271442 | 1 | 0 | BeFree |
| Prostatic Hyperplasia | 0.000271442 | 1 | 0 | BeFree |
| Nasopharyngeal carcinoma | 0.000271442 | 1 | 0 | BeFree |
| Prostatic Neoplasms | 0.000271442 | 1 | 0 | BeFree |
| Malignant tumor of colon | 0.000271442 | 1 | 0 | BeFree |
联系方式
山东省济南市章丘区文博路2号 齐鲁师范学院 genelibs生信实验室
山东省济南市高新区舜华路750号大学科技园北区F座4单元2楼
电话: 0531-88819269
E-mail: product@genelibs.com
微信公众号
关注微信订阅号,实时查看信息,关注医学生物学动态。