ATM (ATM serine/threonine kinase)
- symbol:
- ATM
- locus group:
- protein-coding gene
- location:
- 11q22.3
- gene_family:
- alias symbol:
- TEL1|TELO1
- alias name:
- TEL1, telomere maintenance 1, homo…
- entrez id:
- 472
- ensembl gene id:
- ENSG00000149311
- ucsc gene id:
- uc001pkb.1
- refseq accession:
- NM_000051
- hgnc_id:
- HGNC:795
- approved reserved:
- 1995-07-07
ATM基因(共济失调毛细血管扩张症突变基因)属于PI3K相关激酶(PIKK)家族,该家族成员通常参与细胞周期调控、DNA损伤修复和代谢调节等关键生物学过程。ATM基因编码的蛋白质是一种丝氨酸/苏氨酸蛋白激酶,主要定位于细胞核内,在DNA双链断裂修复中发挥核心作用。当DNA损伤发生时,ATM会被迅速激活,通过磷酸化下游靶蛋白如p53、CHK2和BRCA1等,启动细胞周期检查点阻滞、DNA修复或程序性细胞死亡。ATM基因突变会导致共济失调毛细血管扩张症(AT),这是一种罕见的常染色体隐性遗传病,患者表现为进行性小脑共济失调、免疫缺陷、对电离辐射高度敏感以及癌症易感性增加(特别是白血病和淋巴瘤)。杂合突变携带者也有较高的乳腺癌风险。ATM蛋白功能丧失会导致基因组不稳定性增加,因为细胞无法正确修复DNA损伤或启动凋亡程序。当ATM过表达时,可能增强细胞对DNA损伤的应激反应,但过度激活可能引起不必要的细胞周期阻滞或凋亡;而表达降低则会导致DNA修复缺陷,增加突变积累和癌变风险。除了经典的DNA损伤反应,ATM还参与调控氧化应激反应、端粒维持和自噬过程。在癌症治疗中,ATM状态常被作为预测放疗和某些化疗药物敏感性的生物标志物。PIKK家族的其他成员如ATR和DNA-PKcs也参与DNA损伤应答,但各自识别不同类型的DNA损伤并激活特异的信号通路。ATM特别针对双链断裂,而ATR主要应对单链断裂和复制压力。这个激酶家族的共同特点是都含有C端的激酶结构域,能够磷酸化大量下游底物来协调复杂的细胞应激反应网络。
The protein encoded by this gene belongs to the PI3/PI4-kinase family. This protein is an important cell cycle checkpoint kinase that phosphorylates; thus, it functions as a regulator of a wide variety of downstream proteins, including tumor suppressor proteins p53 and BRCA1, checkpoint kinase CHK2, checkpoint proteins RAD17 and RAD9, and DNA repair protein NBS1. This protein and the closely related kinase ATR are thought to be master controllers of cell cycle checkpoint signaling pathways that are required for cell response to DNA damage and for genome stability. Mutations in this gene are associated with ataxia telangiectasia, an autosomal recessive disorder. [provided by RefSeq, Aug 2010]
由该基因编码的蛋白质属于PI3 / PI4激酶家族。这种蛋白质是一种重要的细胞周期关卡激酶磷酸化;因此,它用作各种各样下游蛋白质,包括肿瘤抑制蛋白p53和BRCA1,检查点激酶CHK2,关卡蛋白RAD17和RAD9,和DNA修复蛋白NBS1的调节器。这种蛋白和紧密相关的激酶的ATR被认为是信令所需要的细胞响应于DNA损伤和基因组稳定性的途径细胞周期关卡的主控制器。在这种基因突变与共济失调毛细血管扩张症,常染色体隐性病症相关。 [由RefSeq的,2010年8月提供]
基因本体信息
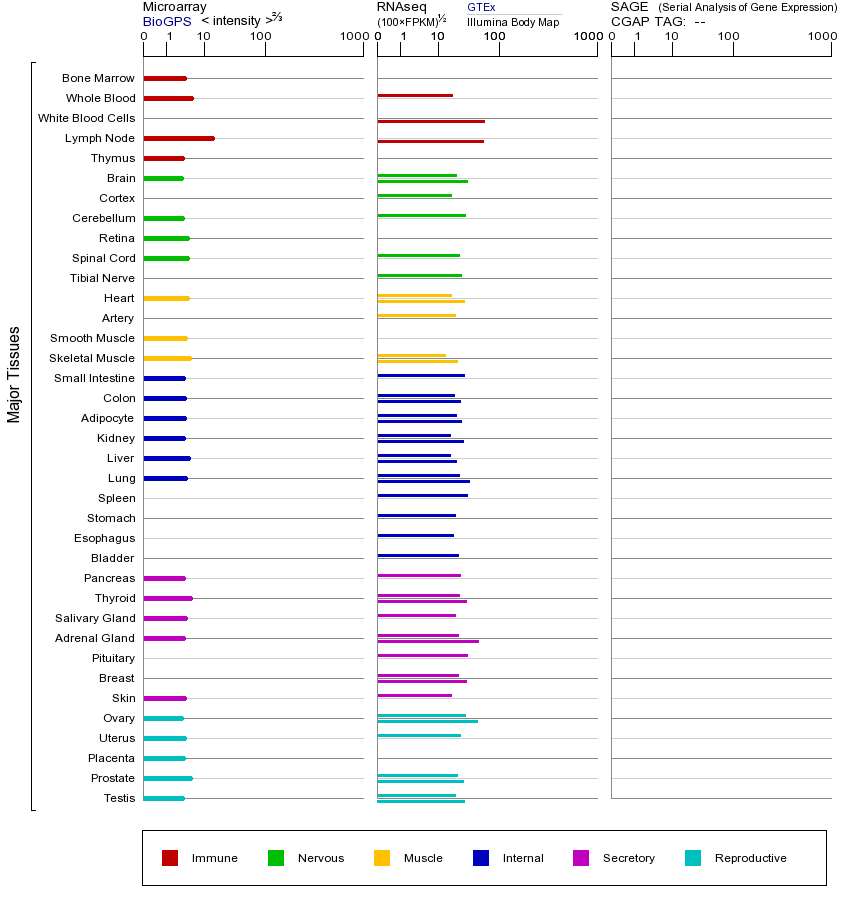
ATM基因(以及对应的蛋白质)的细胞分布位置:
- 质膜
- 细胞质
- 细胞外
- 高尔基体
- 囊泡
- 细胞骨架
- 内质网
- 细胞核
- 内体
- 溶酶体
- 线粒体
ATM基因的本体(GO)信息:
| 名称 |
|---|
| 4064 NF-kappa B signaling pathway [PATH:hsa04064] |
| 4068 FoxO signaling pathway [PATH:hsa04068] |
| 4110 Cell cycle [PATH:hsa04110] |
| 4210 Apoptosis [PATH:hsa04210] |
| 4115 p53 signaling pathway [PATH:hsa04115] |
| 5202 Transcriptional misregulation in cancers [PATH:hsa05202] |
| 5206 MicroRNAs in cancer [PATH:hsa05206] |
| 5166 HTLV-I infection [PATH:hsa05166] |
| 名称 |
|---|
| ATM mediated phosphorylation of repair proteins |
| ATM mediated response to DNA double-strand break |
| Autodegradation of the E3 ubiquitin ligase COP1 |
| Cell Cycle |
| Cell Cycle Checkpoints |
| Cellular response to heat stress |
| Cellular responses to stress |
| Cellular Senescence |
| DNA Damage/Telomere Stress Induced Senescence |
| DNA Repair |
| Double-Strand Break Repair |
| Fanconi Anemia pathway |
| G1/S DNA Damage Checkpoints |
| G2/M Checkpoints |
| G2/M DNA damage checkpoint |
| Homologous Recombination Repair |
| Homologous recombination repair of replication-independent double-strand breaks |
| Meiosis |
| Meiotic recombination |
| p53-Dependent G1 DNA Damage Response |
| p53-Dependent G1/S DNA damage checkpoint |
| p53-Independent DNA Damage Response |
| p53-Independent G1/S DNA damage checkpoint |
| Recruitment of repair and signaling proteins to double-strand breaks |
| Regulation of HSF1-mediated heat shock response |
| Regulation of the Fanconi anemia pathway |
| Stabilization of p53 |
| Ubiquitin Mediated Degradation of Phosphorylated Cdc25A |
| 疾病名称 | 关系值 | NofPmids | NofSnps | 来源 |
| Ataxia Telangiectasia | 0.708689197 | 388 | 49 | BeFree_CLINVAR_CTD_human_GAD_LHGDN_MGD_ORPHANET_UNIPROT |
| Chronic Lymphocytic Leukemia | 0.275089403 | 76 | 0 | BeFree_CTD_human_GAD_LHGDN_ORPHANET |
| Malignant lymphoma, lymphocytic, intermediate differentiation, diffuse | 0.246243163 | 23 | 3 | BeFree_CLINVAR_ORPHANET |
| melanoma | 0.245895776 | 14 | 2 | BeFree_CTD_human_GAD_GWASCAT |
| Mammary Neoplasms | 0.206554329 | 43 | 0 | BeFree_CTD_human_GAD_LHGDN |
| Prostatic Neoplasms | 0.128087174 | 5 | 0 | BeFree_CTD_human_GAD_LHGDN |
| Malignant neoplasm of breast | 0.127502328 | 190 | 9 | BeFree_GAD |
| Pancreatic Neoplasm | 0.127372538 | 6 | 0 | BeFree_CTD_human_GAD |
| T-Cell Prolymphocytic Leukemia | 0.125700279 | 21 | 3 | BeFree_CLINVAR |
| Fibrosis | 0.124734064 | 3 | 0 | CTD_human_GAD |
联系方式
山东省济南市章丘区文博路2号 齐鲁师范学院 genelibs生信实验室
山东省济南市高新区舜华路750号大学科技园北区F座4单元2楼
电话: 0531-88819269
E-mail: product@genelibs.com
微信公众号
关注微信订阅号,实时查看信息,关注医学生物学动态。