GO ( Gene Ontology,基因本体论) 数据库将基因功能分为三大类:分子功能(Molecular Function),生物学过程 (Biological Process) 和 细胞组分 (Cellular Component)。在每一个分类中,都提供一个描述功能信息的分级结构。GO中每一个分类术语都以一种被称为定向非环状图(DAGs)的结构组织起来。研究者可以通过GO分类号和各种GO数据库相关分析工具将分类与具体基因联系起来,从而对这个基因的功能进行描述。研究者可以根据挑选出的差异基因,计算这些差异基因同GO 分类中某(几)个特定的分支的超几何分布关系,GO 分析会对每个有差异基因存在的GO 返回一个p-value,小的p 值表示差异基因在该GO 中出现了富集。GO 分析对实验结果有提示的作用,通过差异基因的GO 分析,可以找到富集差异基因的GO分类条目,寻找不同样品的差异基因可能和哪些基因功能的改变有关。
关联推断分析法采用基因网络和一组注释(GO功能注释)作为输入,并输出每个注释集合的预测得分。关联推断(guilt-by-association)认为:具有共享功能(即,在相同GO注释集合内)的基因是优先的连接的,使用网络中基因关联信息和注释集中的基因信息推断隐藏基因(不在该GO注释集合内)的身份,预测基因网络的关键基因功能(GO)。
数据要求:
1、必须有对照组
2、输入的为基因表达谱数据(gene symbol)
操作步骤(以GEO数据库为例):
1、数据选择:根据疾病或药物选择合适的数据(control,平台,样本量,processed及数据库等)
2、数据本地化:在基因云馆中选择“数据库”,然后选择数据对应的数据库,输入数
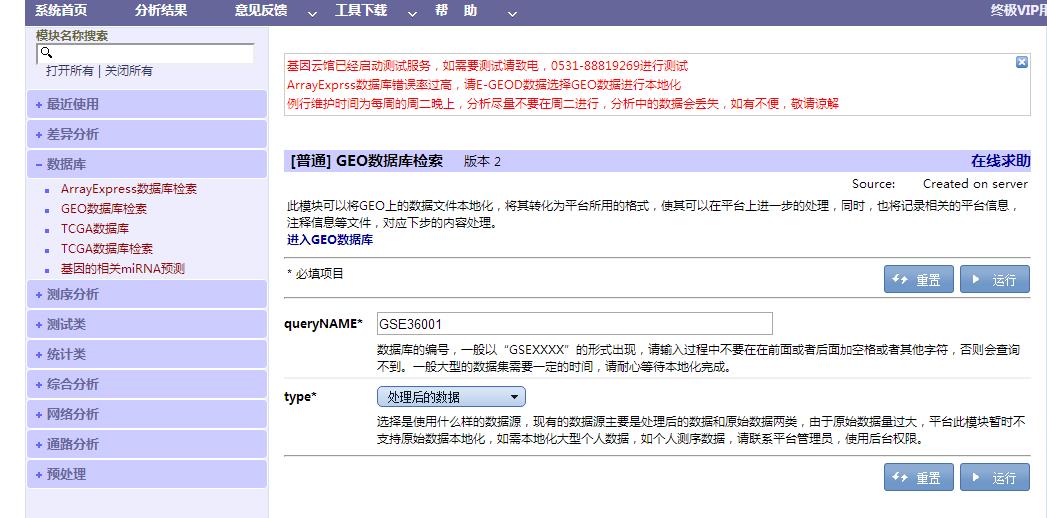
图一:数据本地化处理
3、数据预处理:点击“预处理”→“芯片数据预处理”,eSetPath*处拖入本地化中的RData文件呢,platform出选择数据对应平台,groupName出输入分组信息的列名称(可在本地化的PData文件中找到)。
如果数据的分组信息需要更改,则应先选择“预处理”→“表达集数据信息”输入本地化中的RData文件,运行完成后,更改pDatamatrix.csv内内容(将分组列名称改为groups,如需更改列内对照组、实验组名称则一并更改)然后保存。选择“基因表达集生成器”,“matrix”处输入上步的exprsmatrix.csv文件,“pData”处输入经更改的pDatamatrix.csv,点击运行。如数据分组较多,选择“表达集取子集”后,进行“芯片数据预处理”,若无需取子集,则直接进行“芯片数据预处理”即可。具体操作步骤如下:
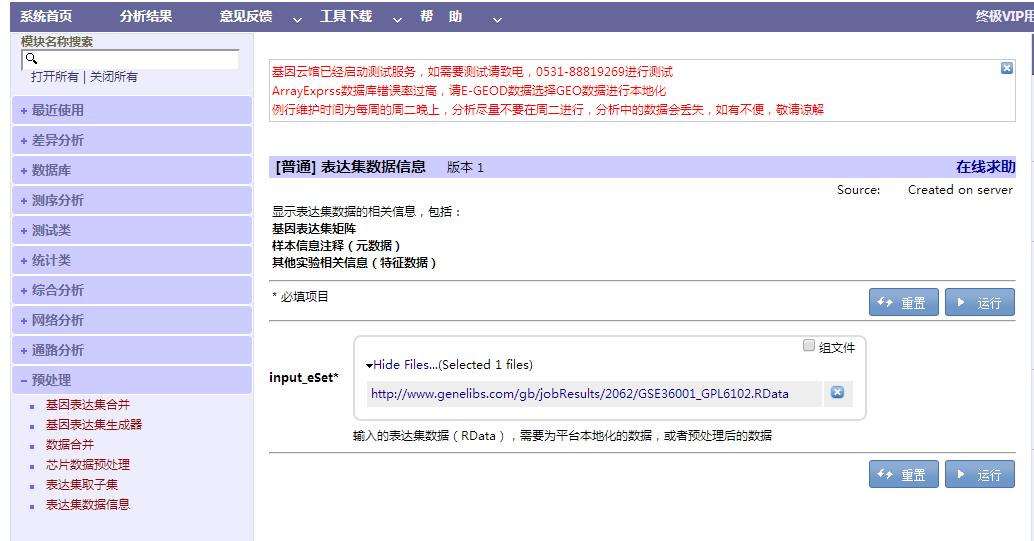
图二:表达集数据信息
注:将此环节得到的“pDatamatrix.csv”文件内容根据需要进行更改(如分组列名称改为groups及组别名称等)并保存
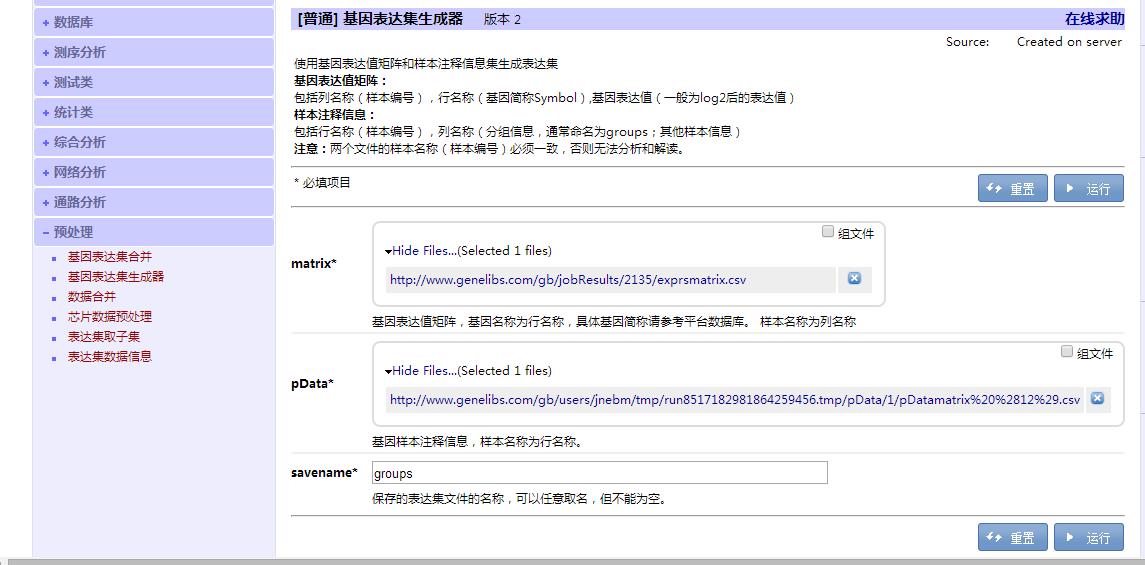
图三:基因表达集生成器
注:此处的pData为更改后的pDatamatrix.csv文件
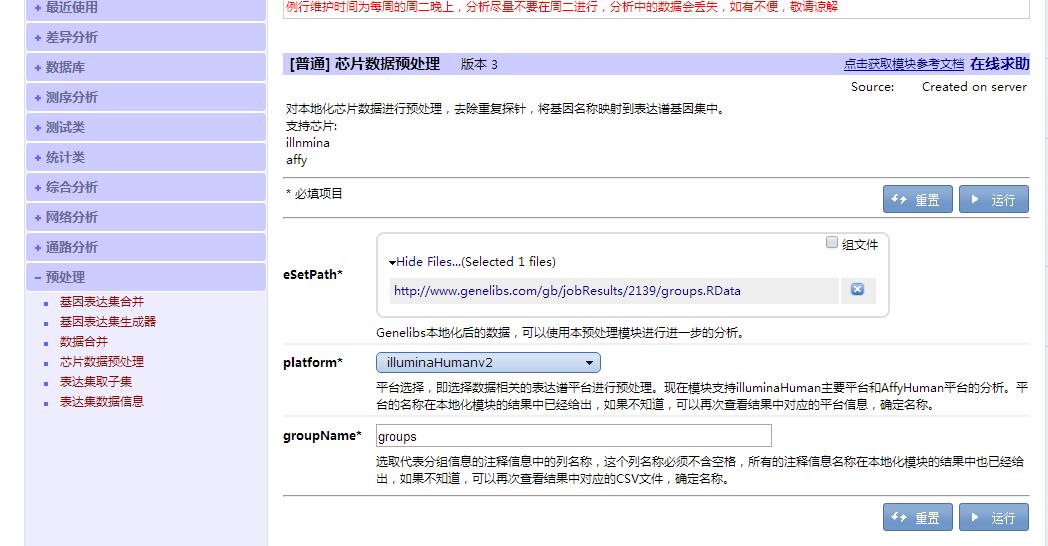
图四:芯片数据预处理
网络关联推断拓展GO分析:选择“网络分析”→“网络关联推断拓展GO分析”,输入预处理后的RData文件及相关参数点击运行即可。
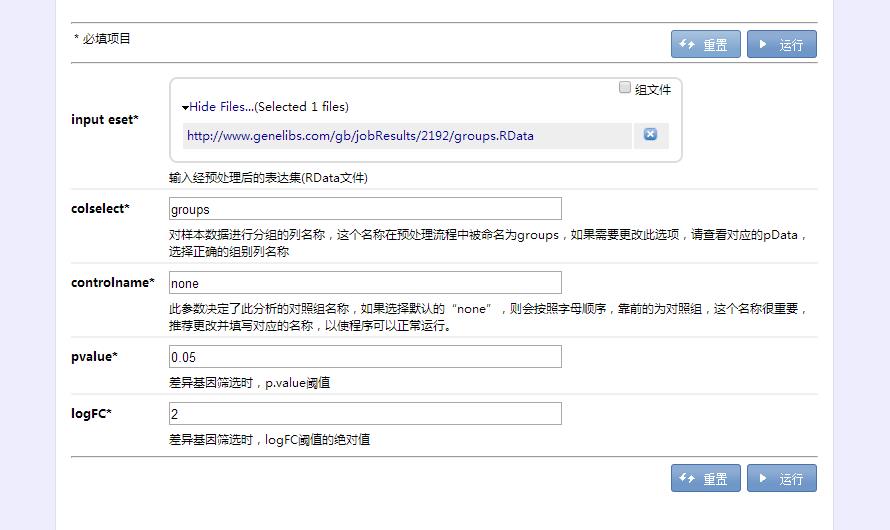
图五:网络关联推断拓展GO分析
结果示例

微信公众号
关注微信订阅号,实时查看信息,关注医学生物学动态。


