Fastq格式详解
FASTQ是基于文本的,保存生物序列(通常是核酸序列)和其测序质量信息的标准格式。其序列以及质量信息都是使用一个ASCII字符标示,最初由Sanger开发,目的是将FASTA序列与质量数据放到一起,目前已经成为高通量测序结果的事实标准。
格式说明
FASTQ文件中每个序列通常有四行:
序列标识以及相关的描述信息,以‘@’开头;
第二行是序列
第三行以‘+’开头,后面是序列标示符、描述信息,或者什么也不加
第四行,是质量信息,和第二行的序列相对应,每一个序列都有一个质量评分,根据评分体系的不同,每个字符的含义表示的数字也不相同。
例如:
@SEQ_ID GATTTGGGGTTCAAAGCAGTATCGATCAAATAGTAAATCCATTTGTTCAACTCACAGTTT + !''*((((***+))%%%++)(%%%%).1***-+*''))**55CCF>>>>>>CCCCCCC65
ILLUMINA SEQUENCE IDENTIFIERS
@HWUSI-EAS100R:6:73:941:1973#0/1
| HWUSI-EAS100R | the unique instrument name |
|---|---|
| 6 | flowcell lane |
| 73 | tile number within the flowcell lane |
| 941 | ‘x’-coordinate of the cluster within the tile |
| 1973 | ‘y’-coordinate of the cluster within the tile |
| #0 | index number for a multiplexed sample (0 for no indexing) |
| /1 | the member of a pair, /1 or /2 (paired-end or mate-pair reads only) |
@EAS139:136:FC706VJ:2:2104:15343:197393 1:Y:18:ATCACG
| EAS139 | the unique instrument name |
|---|---|
| 136 | the run id |
| FC706VJ | the flowcell id |
| 2 | flowcell lane |
| 2104 | tile number within the flowcell lane |
| 15343 | ‘x’-coordinate of the cluster within the tile |
| 197393 | ‘y’-coordinate of the cluster within the tile |
| 1 | the member of a pair, 1 or 2 (paired-end or mate-pair reads only) |
| Y | Y if the read fails filter (read is bad), N otherwise |
| 18 | 0 when none of the control bits are on, otherwise it is an even number |
| ATCACG | index sequence |
NCBI SEQUENCE READ ARCHIVE
@SRR001666.1 071112_SLXA-EAS1_s_7:5:1:817:345 length=36 GGGTGATGGCCGCTGCCGATGGCGTCAAATCCCACC +SRR001666.1 071112_SLXA-EAS1_s_7:5:1:817:345 length=36 IIIIIIIIIIIIIIIIIIIIIIIIIIIIII9IG9IC
关于质量编码格式
质量评分指的是一个碱基的错误概率的对数值。其最初在Phred拼接软件中定义与使用,其后在许多软件中得到使用。其质量得分与错误概率的对应关系见下表:
| PHRED QUALITY SCORE | PROBABILITY OF INCORRECT BASE CALL | BASE CALL ACCURACY |
|---|---|---|
| 10 | 1 in 10 | 90 % |
| 20 | 1 in 100 | 99 % |
| 30 | 1 in 1000 | 99.9 % |
| 40 | 1 in 10000 | 99.99 % |
| 50 | 1 in 100000 | 99.999 % |
Phred quality scores Q are defined as a property which is logarithmically related to the base-calling error probabilities P.
除了Phred质量得分换算标准,还有就是Solexa标准:![]()
两种换算标准的比较:
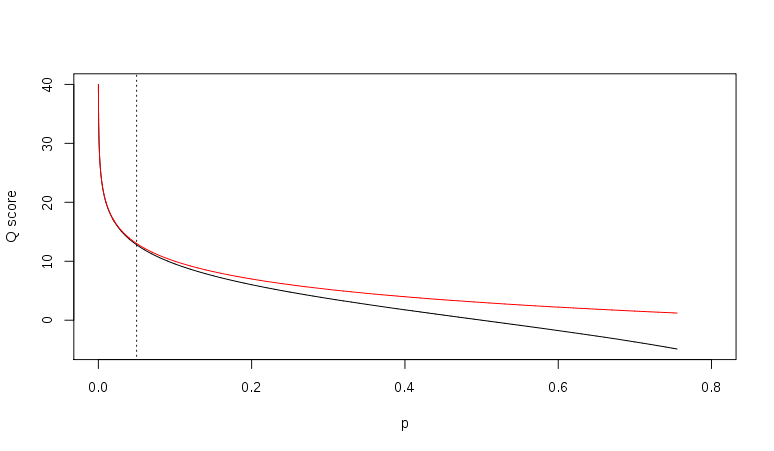
Relationship between Q and p using the Sanger (red) and Solexa (black) equations (described above). The vertical dotted line indicates p = 0.05, or equivalently, Q ≈ 13.
对于每个碱基的质量编码标示,不同的软件采用不同的方案,目前有5种方案:
Sanger,Phred quality score,值的范围从0到92,对应的ASCII码从33到126,但是对于测序数据(raw read data)质量得分通常小于60,序列拼接或者mapping可能用到更大的分数。
Solexa/Illumina 1.0, Solexa/Illumina quality score,值的范围从-5到63,对应的ASCII码从59到126,对于测序数据,得分一般在-5到40之间;
Illumina 1.3+,Phred quality score,值的范围从0到62对应的ASCII码从64到126,低于测序数据,得分在0到40之间;
Illumina 1.5+,Phred quality score,但是0到2作为另外的标示,详见http://solexaqa.sourceforge.net/questions.htm#illumina
Illumina 1.8+
下面是更为直观的表示:
SSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSS.....................................................
..........................XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX......................
...............................IIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIII......................
.................................JJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJ......................
LLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLL....................................................
!"#$%&'()*+,-./0123456789:;?@ABCDEFGHIJKLMNOPQRSTUVWXYZ[]^_`abcdefghijklmnopqrstuvwxyz{|}~
| | | | | |
33 59 64 73 104 126
S - Sanger Phred+33, raw reads typically (0, 40)
X - Solexa Solexa+64, raw reads typically (-5, 40)
I - Illumina 1.3+ Phred+64, raw reads typically (0, 40)
J - Illumina 1.5+ Phred+64, raw reads typically (3, 40)
with 0=unused, 1=unused, 2=Read Segment Quality Control Indicator (bold)
(Note: See discussion above).
L - Illumina 1.8+ Phred+33, raw reads typically (0, 41)文件后缀
没有特别的规定,通常使用.fq, .fastq, .txt等。
格式转换:
Biopython version 1.51 onwards (interconverts Sanger, Solexa and Illumina 1.3+)
EMBOSS version 6.1.0 patch 1 onwards (interconverts Sanger, Solexa and Illumina 1.3+)
BioPerl version 1.6.1 onwards (interconverts Sanger, Solexa and Illumina 1.3+)
BioRuby version 1.4.0 onwards (interconverts Sanger, Solexa and Illumina 1.3+)
BioJava version 1.7.1 to 1.8.x (interconverts Sanger, Solexa and Illumina 1.3+)
MAQ can convert from Solexa to Sanger (use this patch to support Illumina 1.3+ files).
fastx_toolkit The included fastq_quality_converter program can convert Illumina to Sanger
参考以及相关链接
Cock et al (2009) The Sanger FASTQ file format for sequences with quality scores, and the Solexa/Illumina FASTQ variants. Nucleic Acids Research, doi:10.1093/nar/gkp1137
MAQ webpage discussing FASTQ variants
Fastx toolkit collection of command line tools for Short-Reads FASTA/FASTQ files preprocessing
Fastqc quality control tool for high throughput sequence data
联系方式
山东省济南市章丘区文博路2号 齐鲁师范学院 genelibs生信实验室
山东省济南市高新区舜华路750号大学科技园北区F座4单元2楼
电话: 0531-88819269
E-mail: product@genelibs.com
微信公众号
关注微信订阅号,实时查看信息,关注医学生物学动态。


