现有的分析列表:
| 名称 (版本) | 模块信息和描述文档 |
| ArrayExpress数据库检索 (1) | 此模块可以将ArrayExprss上的数据文件本地化,将其转化为平台所用的格式,使其可以在平台上进一步的处理, 同时,也将记录相关的平台信息,注释信息等文件,对应下步的内容处理。 [no documentation] |
| GEO数据库检索 (2) | 此模块可以将GEO上的数据文件本地化,将其转化为平台所用的格式,使其可以在平台上进一步的处理,同时,也将记录相关的平台信息,注释信息等文件,对应下步的内容处理。 [no documentation] |
| KEGG通路图谱 (1) | 使用pathview技术结合KEGG通路数据库以及倍增关系检测,进行通路的可视化分析。 [no documentation] |
| TCGA数据库检索 (2) | 生成TCGA数据库的本地化数据,进行标准化和预处理,使之可以进行后续的分析。 [no documentation] |
| 个性化通路分析 (1.2) | 通过算法,将个体的通路变化计算出来,得到一个对应的个体化图谱。 [no documentation] |
| 中文名称的模块 (2) | 中文名称 [no documentation] |
| 基因网络模块熵值 (4) | 根据STRING数据库鉴别疾病在两种条件下的相似或相同的模块,并利用熵值计算筛选上述模块中的差异模块。 [no documentation] |
| 基因表达集生成器 (2) | 使用基因表达值矩阵和样本注释信息集生成表达集 基因表达值矩阵: 包括列名称(样本编号),行名称(基因简称Symbol),基因表达值(一般为log2后的表达值) 样本注释信息: 包括行名称(样本编号),列名称(分组信息,通常命名为groups;其他样本信息) 注意:两个文件的样本名称(样本编号)必须一致,否则无法分析和解读。 |
| 差异基因与差异通路分析 (1) | 通过LIMMA法,鉴定疾病的差异基因,通过吸引子算法,计算疾病的差异通路,并进行差异基因的火山图,倍增关系图,目的基因差异箱式图的绘制。同时将结果以表格的形式进行存储 [no documentation] |
| 差异基因分析 (1) | 通过LIMMA法,鉴定疾病的差异基因,并进行差异基因的火山图,倍增关系图,目的基因差异箱式图的绘制。同时将结果以表格的形式进行存储。 readme.txt |
| 差异基因网络通路分析 (7) | 本流程是测试流程,流程思路为将表达谱数据进行差异基因分析,网络分析以及通路富集分析,并在线生成相应的报告。 [no documentation] |
| 疾病子网络分析 (2) | 目的:因研究样本量小及基因表达测量的高噪声干扰,要从基因表达谱中挖掘出新的生物标记基因对疾病进行精确分类非常困难。早期的很多研究都提出了通过集成基因表达谱和蛋白质相互作用网络分析方法,找到诊断子网络的标记基因的方法。 然而,这些方法并没有充分考虑到网络中基因之间的邻里关系,不能识别许多潜在的标记基因。本研究的主要思想是充分利用生物学拓扑属性,观测那些与相同或相似的疾病有相互联系的基因,这些基因通常存在于相同的分子网络的周围。 本研究提出了一个基于自我中心网络分析技术的分析方法,从大规模的生物网络中详尽搜索并按优先顺序区分出疾病子网络和标记基因。最后将区分出的疾病子网络进行通路富集分析。 分析简图: 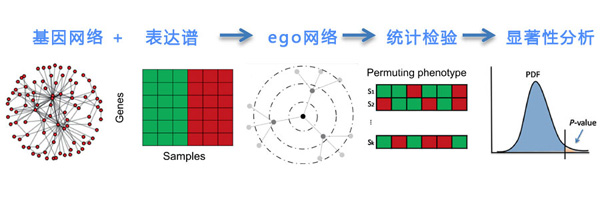 [no documentation] |
| 芯片数据预处理 (3) | 对本地化芯片数据进行预处理,去除重复探针,将基因名称映射到表达谱基因集中。 支持芯片: illnmina affy readme.txt |
| 表达集取子集 (3) | 一个表达集往往有多组,例如多种药物处理,多种实验设计,疾病分期等等。需要将表达集中的合适数据去除,例如留下所需的两组,进行后续比较,因此,需要使用取子集模块,取得所需的子集进行操作。 [no documentation] |
| 表达集数据信息 (1) | 显示表达集数据的相关信息,包括: 基因表达集矩阵 样本信息注释(元数据) 其他实验相关信息(特征数据) [no documentation] |
| 通路药物分析(CMAP药物) (2) | 使用cMAP药物数据库,检测5千余种药物对细胞全基因组表达变化的影响,建立药物对通路影响的数据库。并使用spearman相关系数检测疾病通路数据与药物的相关性。正相关表示药物和疾病影响趋势相同,负相关表示疾病影响与药物趋势相反。 [no documentation] |
微信公众号
关注微信订阅号,实时查看信息,关注医学生物学动态。


